

背景介绍
你敢信吗?以前在显微镜下挤成“一团乱麻”的细菌mRNA,如今可被精准分离并逐一分析。哈佛团队最新研发的Bacterial-MERFISH技术,通过将细菌体积物理膨胀1000倍,结合多重容错性荧光原位杂交(MERFISH)技术,精准定位每个基因的表达位置,这项登上Science的黑科技,彻底打破了微生物空间研究的天花板!

微生物研究的“空间困境”
为何传统方法难窥全貌?
做微生物研究的伙伴肯定深有体会:细菌体积仅为真核细胞的几十分之一,而内部mRNA密度极高。一个大肠杆菌细胞内约包含8000个mRNA分子,传统荧光原位杂交(FISH)根本分不清谁是谁;就算用单细胞测序,也得把细菌从生物膜或宿主组织中分离,宝贵的空间位置信息全丢了。
1000倍膨胀+MERFISH
重构细菌研究范式
Bacterial-MERFISH 技术,说起来很直观,既然细菌太小、mRNA太挤,那就把它放大!
研究团队为细菌量身定制了一套膨胀方案:先把细菌用多聚甲醛(PFA)固定,消化掉坚硬的细胞壁,加水让凝胶膨胀,单轮膨胀就能让细菌体积变大50倍,两轮迭代下来体积直接扩到1000倍!原本挤在一起的mRNA分子,瞬间被拉开距离,再结合MERFISH技术的组合荧光探针,就像给每个mRNA发“专属身份证”,精准识别数千种不同的转录本。
该技术的核心性能表现优异:在大肠杆菌里能同时分析1930个操纵子,覆盖80%的转录组,检测效率最高达50%,假阳性率还极低,能够跨4个数量级精准量化基因表达水平。
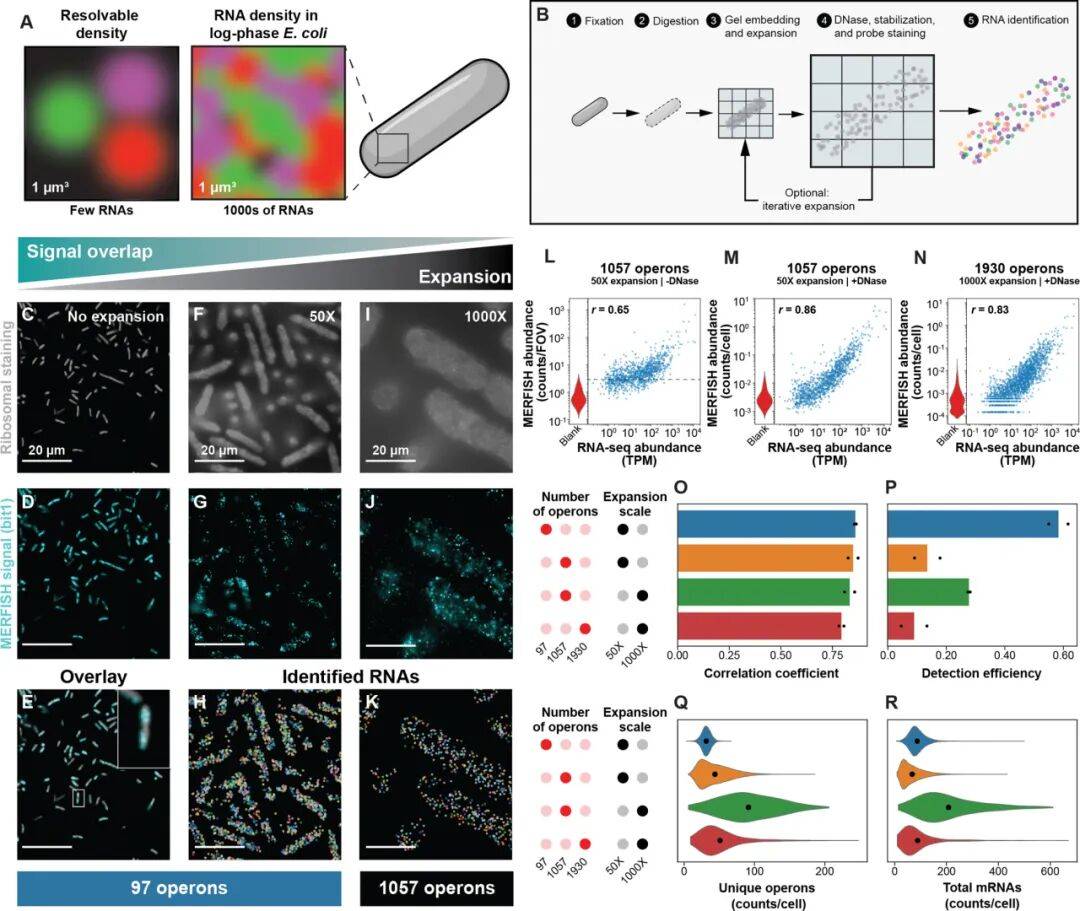
Fig1 大肠杆菌中数千个操纵子的图像表达分析
三大突破性发现
重新认识细菌
有了Bacterial-MERFISH这个工具,研究团队取得了多项颠覆性发现:
01 细菌应对碳饥饿
不是“同步行动”,而是“分层探索”
当大肠杆菌从葡萄糖切换到木糖代谢时,传统研究认为菌群会同步调整代谢模式。但通过Bacterial-MERFISH观察发现,细菌会分化为十几个功能亚群,采取差异化策略:部分亚群先动员糖原储备,部分尝试分解麦芽糖、甘油等碳源,最终才启动木糖和阿拉伯糖代谢途径。这种“分层探索”策略类似细菌的“风险分散机制”,确保种群在环境变化中维持存活,展现了微生物复杂的适应性进化智慧。
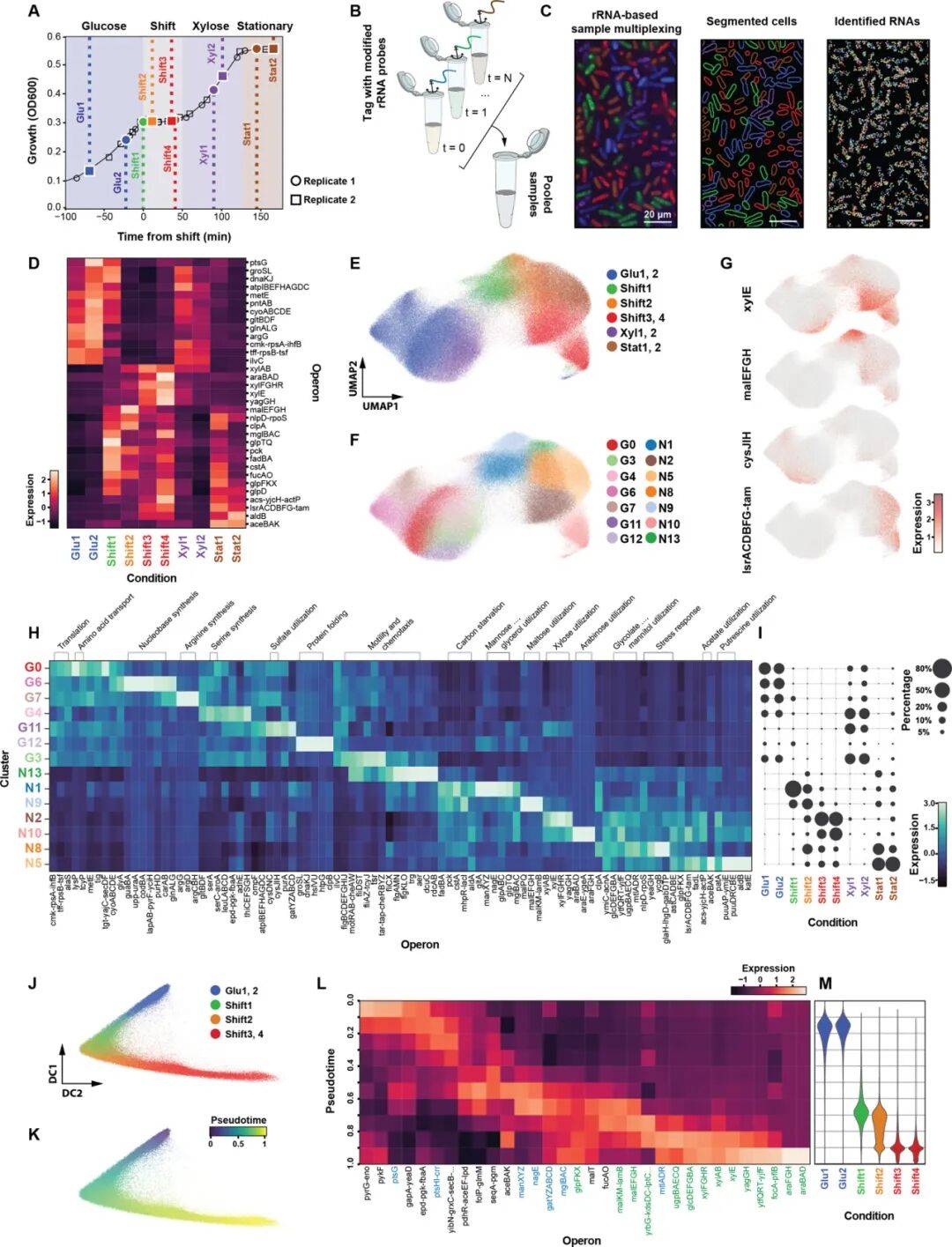
Fig2 碳饥饿的随机分层响应
02 细菌内部不混乱
mRNA有清晰的“空间地图”
长期以来,细菌内部被认为是分子随机分布的“无序系统”,但该研究首次绘制出细菌亚细胞层面的转录组图谱mRNA存在明确的空间分布模式,部分mRNA富集于细胞膜(如编码内膜蛋白的转录本),部分聚集于细胞两极,另有部分定位于细胞中央。更重要的是,mRNA的空间位置与其在染色体上的起源位点密切相关,例如靠近复制终点terC的基因,其转录产物多聚集于细胞中部,这种“基因定位-转录本分布”的关联规律,此前尚未被发现。
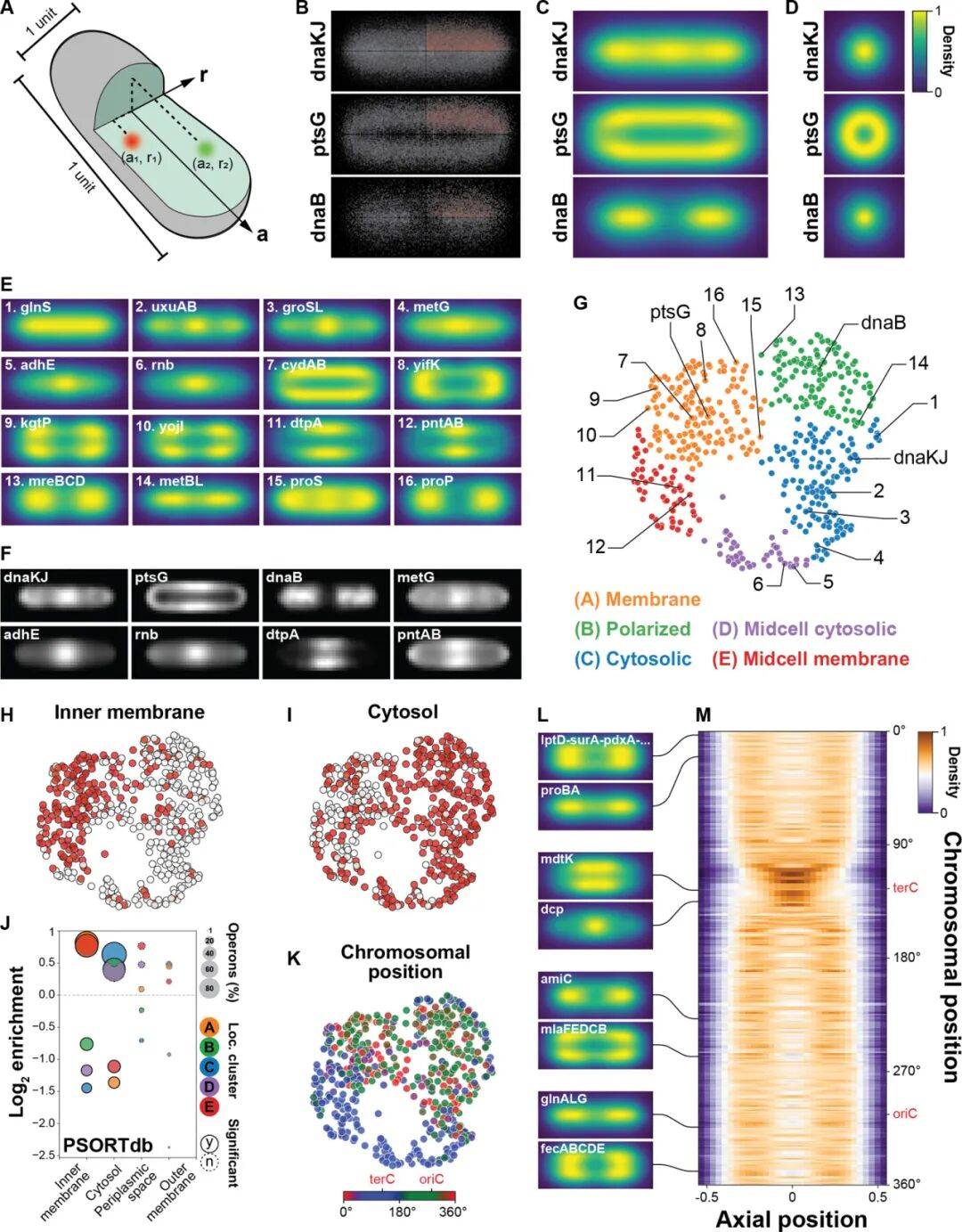
Fig3 大肠杆菌转录组呈现出多样化的模式
03 肠道菌的“微米级适应”
在黏液层和肠腔,代谢完全不一样
研究团队为无菌小鼠定植人类肠道常见的多形拟杆菌后,利用Bacterial-MERFISH技术观察其在结肠内的分布与代谢状态。结果显示,靠近宿主黏液层的细菌会特异性上调宿主聚糖利用相关基因(如靶向黏蛋白的PULs),而位于肠腔中央的细菌则优先表达饮食聚糖分解相关基因。这一发现证实,肠道细菌在微米级空间范围内,已演化出适应局部微环境的特异性代谢策略,这种精细差异在传统批量测序中无法被捕捉。
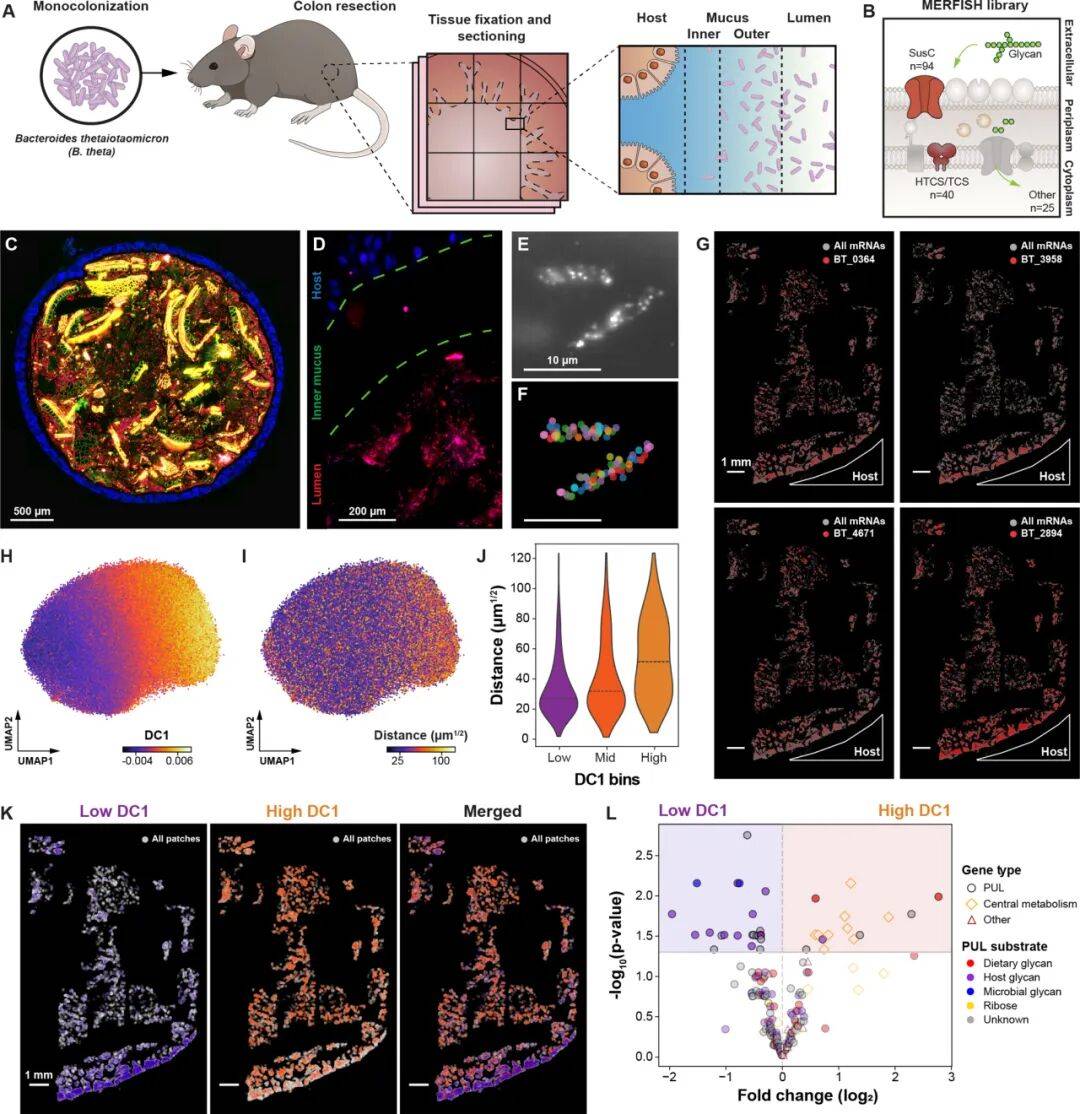
Fig4 肠道共生菌对哺乳动物结肠中微米级生态位的适应
对科研人来说
Bacterial-MERFISH技术意味着什么?
对于微生物领域的科研人员而言,不管是肠道菌群、环境微生物还是病原菌,Bacterial-MERFISH都可能成为你的“科研加速器”:
生物膜研究中
可以直接看到不同细菌亚群的功能分工,无需依赖分离培养推测机制
宿主-微生物互作研究中
能精准定位病原菌在宿主组织中的定植位点,以及不同微环境下的基因表达变化
针对难培养微生物
可直接在原位分析其功能特征,突破传统培养技术的限制。
从“看不清”到“看得透”,从“批量平均”到“单细胞空间精准分析”,Bacterial-MERFISH不仅是一项技术突破,更给微生物研究打开了一扇新大门。未来不管是解析复杂菌群的互作机制,还是开发针对病原菌的精准干预方案,这项技术都可能成为核心工具。
拜谱小结
拜谱生物作为国内领先的多组学技术服务公司,构建了完善成熟的技术服务体系。深度布局单细胞及空间研究领域,可提供全面的真核原核单细胞转录组测序与分析服务,同时涵盖蛋白质组学、修饰组学、代谢组学、高通量测序等核心组学技术,能通过多组学数据整合挖掘与深度机制解析,精准匹配微生物学、肠道生态、细菌功能异质性等领域的研究需求。从技术支持到数据分析全链条赋能,助力科研人员解锁细菌空间调控、微生物互作等关键科学问题,冲刺高分文章!欢迎咨询合作,共同推进科研突破!
参考文献
Sarfatis A, Wang Y, Twumasi-Ankrah N, Moffitt JR. Highly multiplexed spatial transcriptomics in bacteria. Science. 2025 Jan 24;387(6732):eadr0932. doi: 10.1126/science.adr0932.