

单细胞测序技术的爆发,让我们得以窥见细胞群体的“微观多样性”。但拿到海量的单细胞表达矩阵后,最关键的一步便是细胞注释,明确每个细胞的身份,这是后续差异分析、功能富集的基础。
一款合适的自动注释工具能让科研效率翻倍!今天,拜谱生物为大家汇总了8款主流细胞注释工具,涵盖“参考数据集匹配”、“marker基因匹配”、“专属细胞类型注释”三大场景,快收藏起来按需选用。
BIOPROFILE
基于参考数据集的自动注释工具
1、SingleR
经典老牌,参考库匹配的标杆工具
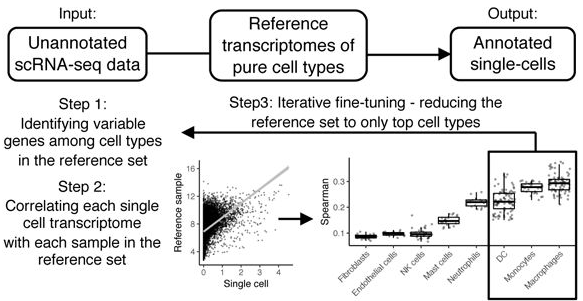
由Dvir Aran研发团队开发的SingleR,是一款基于R语言的单细胞转录组测序数据分析工具,它通过计算待注释单细胞表达谱与参考数据集中各细胞类型Marker的Spearman相关系数实现细胞类型自动注释,并可提供置信度分数以助力识别注释存疑的细胞。SingleR内置了7个参考数据集,涵盖人类和小鼠的多种细胞类型,适用于不同物种及组织的单细胞结果注释。SingleR的突出优势在于操作相对简便,能快速为研究人员提供初步且可靠的细胞类型注释结果,这使其成为当前应用最广泛的工具之一。其注释效果的核心在于为分析任务匹配最合适的参考数据集,当参考数据与待注释样本高度契合时,其稳健的相关系数法能获得极为可靠的全基因组注释。因此,它尤其适合作为分析流程中的核心基础工具,为后续更精细的亚型分析提供坚实框架。
2、ScType
精准度拉满,支持细分细胞亚群
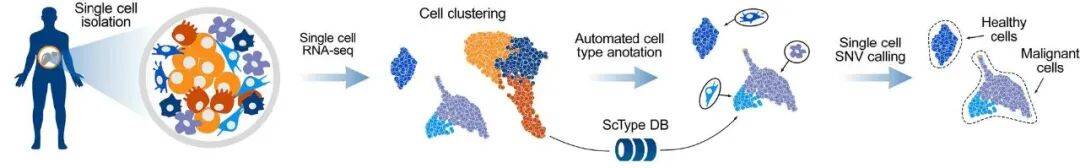
由芬兰赫尔辛基大学分子医学研究所(FIMM)研发的ScType,是一款专注于单细胞转录组数据(scRNA-seq)的全自动细胞类型鉴定工具,以细胞亚型区分精度高、适配免疫细胞或组织特异性亚群精细注释为主要优势。其核心技术在于从scRNA-seq数据中自动识别并筛选特定细胞类型的关联标记基因,通过整合正负标记基因的双重信息生成细胞类型评分矩阵,为每个细胞(或细胞簇)精准匹配最可能的身份,尤其在区分T细胞亚群等高度相似的免疫细胞亚型时表现突出。为提升易用性,ScType不仅提供开源R包(支持Seurat对象直接调用,可通过自定义标记文件输入专属基因集),还搭建了交互式网页平台(https://sctype.app),同时具备自动检测数据集组织类型的功能,助力科研人员更高效地完成细胞类型精确鉴定。
3、clustifyr
灵活适配,跨平台兼容性强
面对单细胞注释中参考数据稀缺或不匹配的挑战,clustifyr提供了高效的解决方案。作为一款功能灵活的单细胞注释R包,clustifyr实现了“数据无关”的注释理念:它允许用户直接使用bulk RNA-seq、scRNA-seq或自定义基因列表作为参考,极大拓展了其应用边界。该工具通过计算待注释数据与参考集之间的斯皮尔曼相关性来分配细胞类型,并可结合多种富集分析策略,确保注释结果的准确性。无论是进行跨平台数据整合,还是针对小众物种或自建数据集的分析,clustifyr 都能提供高度适配的细胞类型鉴定方案。

BIOPROFILE
基于marker基因的自动注释工具
1、SCINA
无参考库也能标,Marker 基因驱动
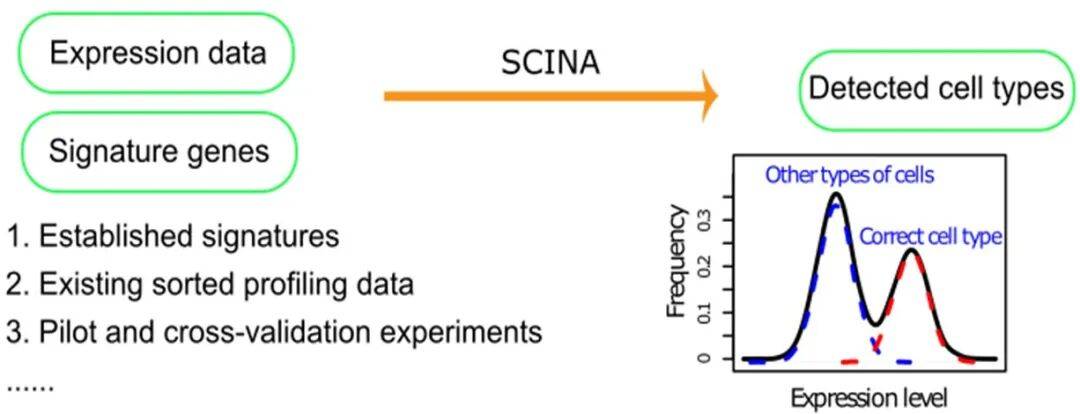
SCINA是一款基于Marker基因的半监督注释工具,其核心能力是“无参考注释”。它利用期望最大化(EM)算法进行迭代计算,为每种细胞类型构建两个高斯分布模型:一个针对其Marker基因的高表达,另一个针对非Marker基因的低表达。这种机制使其能够精准识别出那些同时高表达某一类型Marker、又低表达其他类型基因的“典型细胞”。更重要的是,SCINA支持多标签注释。这意味着当一个细胞同时表达两种及以上细胞类型的特征基因时,它不会被强制归为单一类型,而是被识别为可能处于过渡状态或具有混合特征的细胞,并为每种可能性提供可解释的后验概率。
2、Garnett
可训练的“个性化注释工具”
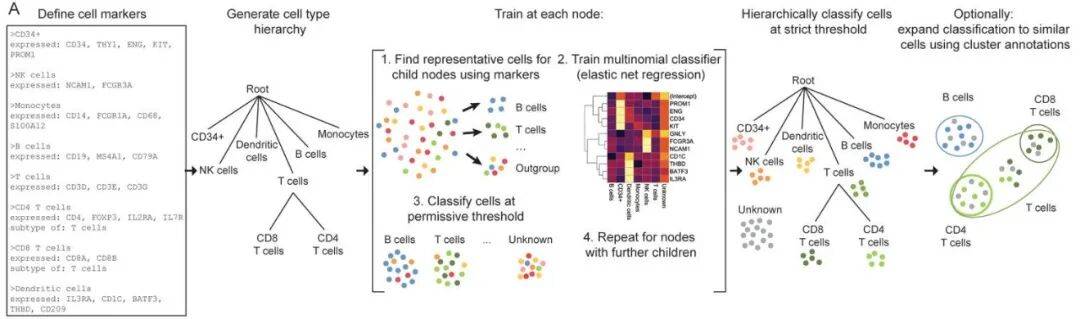
Garnett是一种基于可训练分类器的注释工具。其流程始于用户根据先验知识构建一个标记基因文件,以定义基本的细胞类型。随后,工具会运用弹性网络回归模型,以用户定义的标记为起点,在单细胞数据中进行迭代训练。该模型不仅能自动筛选和优化初始标记基因的权重,更能主动发掘数据中新的、有判别力的基因特征作为补充。最终生成的分类器,是一个融合了专家知识与数据驱动发现的强大模型,确保了注释的准确性与在同类数据集中的可移植性。
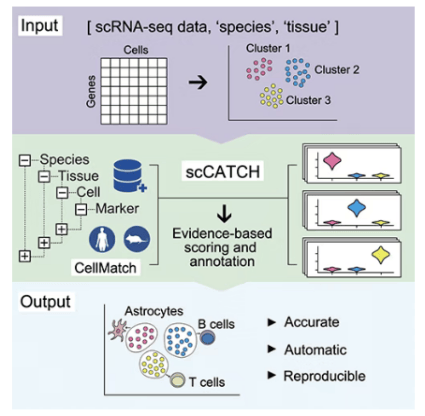
3、scCATCH
细胞类型“知识库”,覆盖广且准
scCATCH通过一种基于证据的、聚类水平的自动化流程进行细胞注释,其过程类似于手动注释。该工具首先识别每个细胞聚类中特异性高表达的基因,然后将这些基因与内置的“CellMatch”知识库进行比对。该知识库整合了来自大量文献的组织特异性细胞类型及其标记基因证据。通过综合评估标记基因在特定组织中的表达特异性、以及其在当前聚类中的表达水平,scCATCH会为每个聚类分配一个或多个潜在的细胞类型,并给出相应的置信分数。由于该方法依赖于标记基因列表而非完整的表达谱,其内置数据集更精简,却能兼容更多的组织和物种。
BIOPROFILE
专属细胞类型自动注释工具
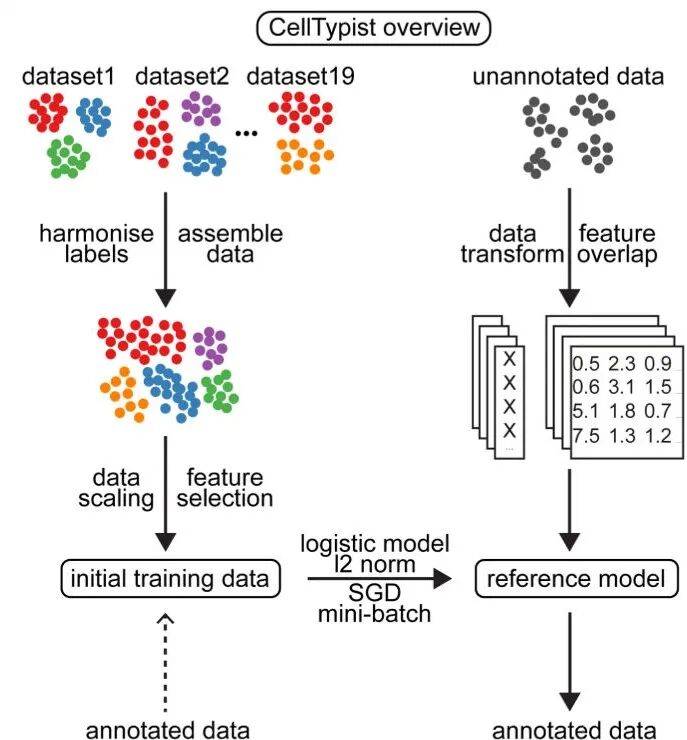
1、CellTypist
免疫细胞注释“专家级”工具
CellTypist是一款专为人体免疫细胞设计的高分辨率自动注释工具。其核心优势源于一个经过精心整理的庞大训练集,该集合成了来自多种人体组织的数百万个免疫细胞。基于此,CellTypist采用逻辑回归模型进行训练,并优化了基因权重,最终生成一个覆盖极其广泛(包括T细胞、B细胞、NK细胞、髓系细胞等)且细分至200余种细胞状态/亚型的分类器。此外,它还包含一项关键的“过半数投票”机制:当提供细胞聚类信息时,工具会统计每个聚类内所有细胞的预测结果,并将占比最高的细胞类型(须超过50%)指定为该聚类的共识标签。这一功能通过整合群体信息,有效提升了最终注释结果的稳健性和可解释性。
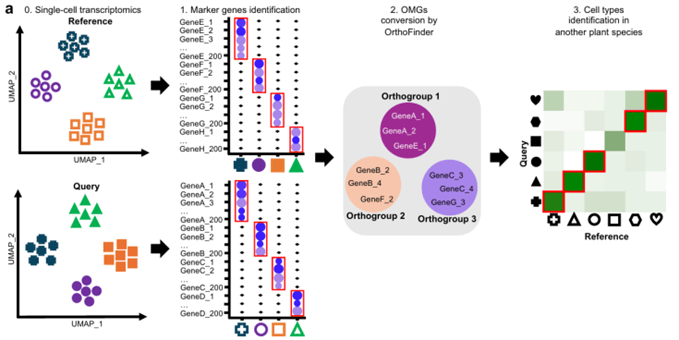
2、OMG Browser
植物单细胞注释“独苗”工具
OMG Browser是当前植物单细胞研究领域的关键工具,由中国科学院遗传与发育生物学研究所主导开发。它解决了植物学研究中的一个核心难题:如何将模式植物(如拟南芥)的细胞类型知识迁移到研究基础薄弱的重要作物(如水稻、玉米)中。其核心方法是构建直系同源标记基因(Orthologous Marker Groups,简称OMGs),这些OMGs是不同物种间保守的、具有相同细胞类型指示功能的基因家族。OMG Browser通过识别目标数据与参考数据中细胞群所共享的OMGs数量,来判断它们是否属于同一种细胞类型。这种方法绕开了因基因名称不统一而造成的比较障碍,为植物的跨物种细胞图谱绘制提供了统一的标准和分析框架。
工具选择指南&结语
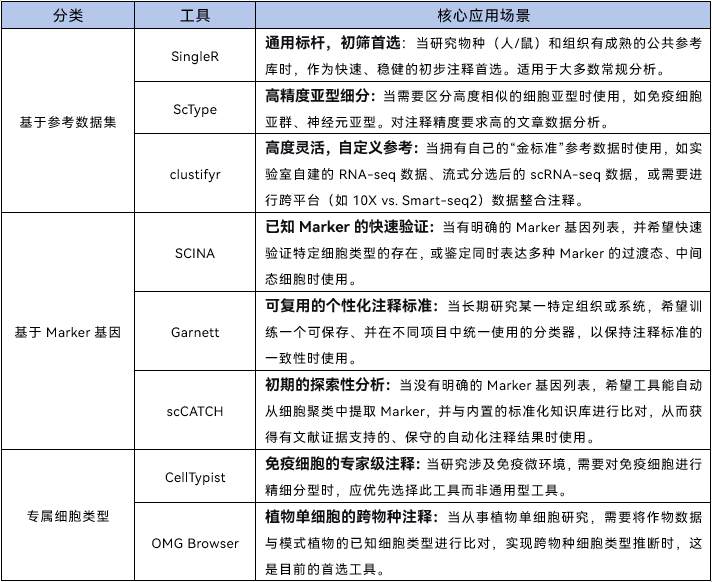
本文汇总的8款工具覆盖了单细胞注释的绝大多数场景,从通用型到专属型,从动物到植物,总有一款适合你的研究!但需注意:工具注释结果仅为“参考”,最终需结合实验验证进行确认。如果您在数据分析中需要专业的技术支持,欢迎随时与我们联系!
全国免费统一热线:400-820-8531
参考文献
[1] Aran D, Looney AP, Liu L, et al. Reference-based analysis of lung single-cell sequencing reveals a transitional profibrotic macrophage. Nat Immunol. 2019 Feb;20(2):163-172. doi: 10.1038/s41590-018-0276-y. Epub 2019 Jan 14. PMID: 30643263; PMCID: PMC6340744.
[2] Ianevski A, Giri AK, Aittokallio T. Fully-automated and ultra-fast cell-type identification using specific marker combinations from single-cell transcriptomic data. Nat Commun. 2022 Mar 10;13(1):1246. doi: 10.1038/s41467-022-28803-w. PMID: 35273156; PMCID: PMC8913782.
[3] Fu R, Gillen AE, Sheridan RM, et al. clustifyr: an R package for automated single-cell RNA sequencing cluster classification. F1000Res. 2020 Apr 1;9:223. doi: 10.12688/f1000research.22969.2. PMID: 32765839; PMCID: PMC7383722.
[4] Zhang Z, Luo D, Zhong X, et al. SCINA: A Semi-Supervised Subtyping Algorithm of Single Cells and Bulk Samples. Genes (Basel). 2019 Jul 12;10(7):531. doi: 10.3390/genes10070531. PMID: 31336988; PMCID: PMC6678337.
[5] Pliner HA, Shendure J, Trapnell C. Supervised classification enables rapid annotation of cell atlases. Nat Methods. 2019 Oct;16(10):983-986. doi: 10.1038/s41592-019-0535-3. Epub 2019 Sep 9. PMID: 31501545; PMCID: PMC6791524.
[6] Shao X, Liao J, Lu X, et al. scCATCH: Automatic Annotation on Cell Types of Clusters from Single-Cell RNA Sequencing Data. iScience. 2020 Mar 27;23(3):100882. doi: 10.1016/j.isci.2020.100882. Epub 2020 Feb 4. PMID: 32062421; PMCID: PMC7031312.
[7] Domínguez Conde C, Xu C, Jarvis LB, et al. Cross-tissue immune cell analysis reveals tissue-specific features in humans. Science. 2022 May 13;376(6594):eabl5197. doi: 10.1126/science.abl5197. Epub 2022 May 13. PMID: 35549406; PMCID: PMC7612735.
[8] Chau TN, Timilsena PR, Bathala SP, et al. Orthologous marker groups reveal broad cell identity conservation across plant single-cell transcriptomes. Nat Commun. 2025 Jan 2;16(1):201. doi: 10.1038/s41467-024-55755-0. PMID: 39747890; PMCID: PMC11695714.