

基因的微小差异如何变成疾病、耐药等复杂表型?这是生命科学领域困扰百年的“基因型-表型鸿沟”—尽管基因组测序已普及,但大多数自然变异功能仍不明确。
2025年10月9日,斯坦福大学Daniel F. Jarosz团队(酵母遗传学顶级团队)与德国柏林夏里特医学院Markus Ralser团队(代谢组学权威)联合在Science上发表了题为“A genome-to-proteome map reveals how natural variants drive proteome diversity and shape fitness”的研究文章。通过整合超高精度蛋白质组与基因组数据,首次绘制了核苷酸分辨率的“基因-蛋白质”全景图谱。这不仅是一次技术飞跃,更为我们理解自身健康、预测疾病风险打开了全新的大门。

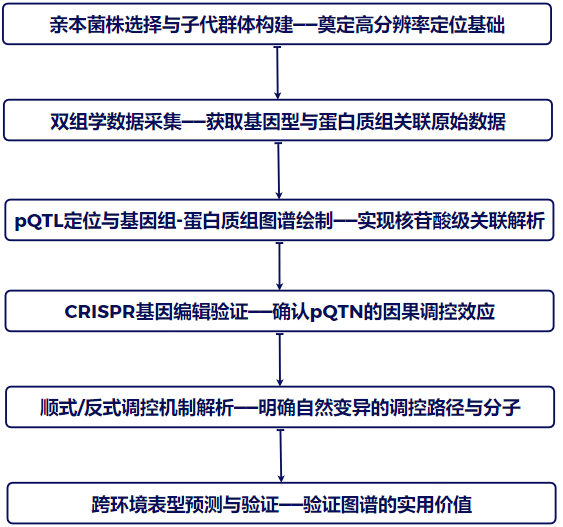
技术路线
研究结果
1.自然遗传变异驱动显著的蛋白质组多样性
研究发现两种亲本酵母(葡萄园菌株RM、临床菌株YJM)仅存在~0.1%的遗传差异,但在蛋白质水平上,1225个检测蛋白中有826个(67.4%)表现出显著表达差异,且差异蛋白具有明确的功能相关性—YJM菌株中氨基酸合成、嘌呤合成及糖异生相关蛋白丰度更高,RM菌株中氧化磷酸化和三羧酸循环(TCA)相关蛋白丰度更高(图1B-D)。
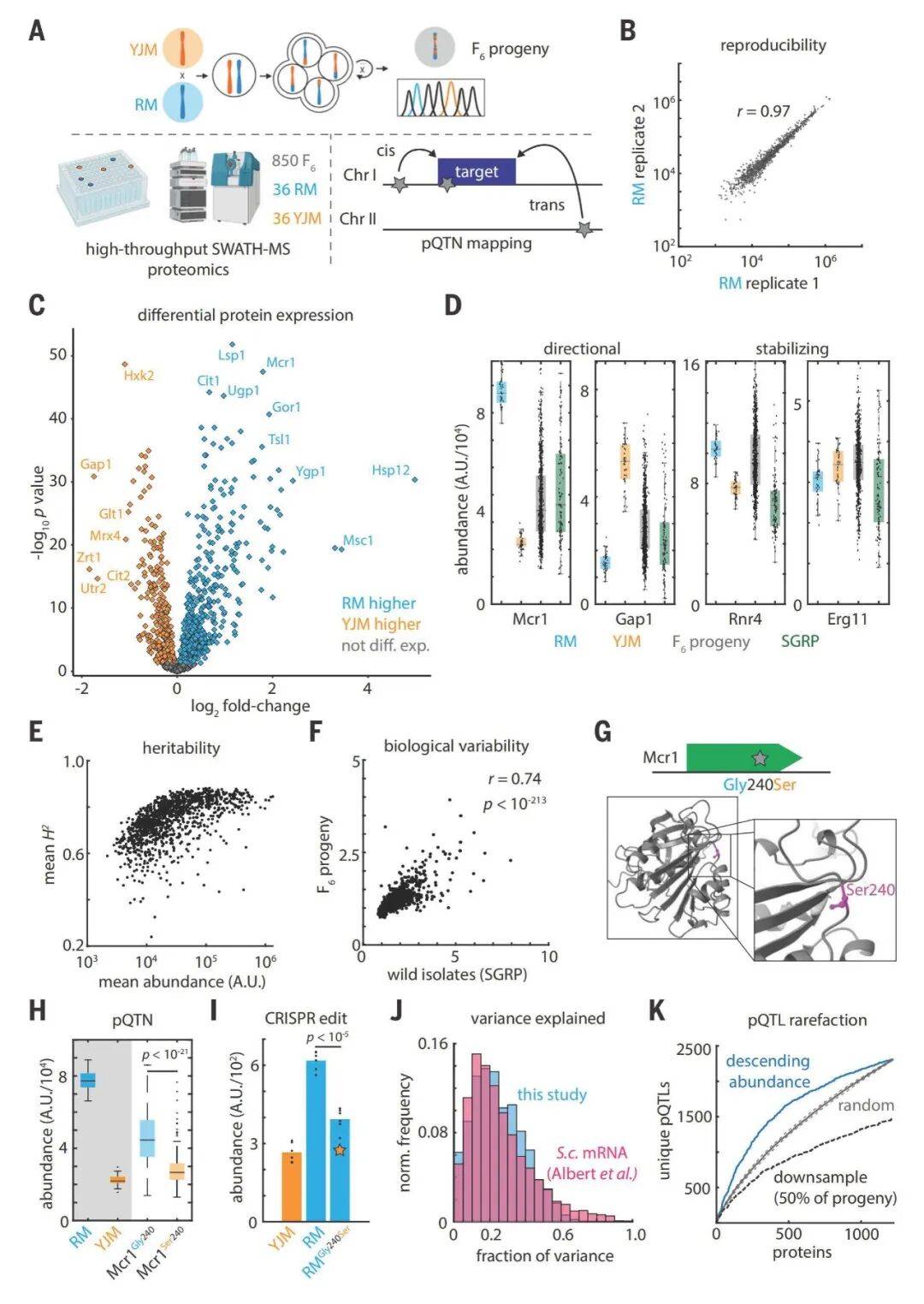 图1基因组到蛋白质组全景图谱的构建流程与关键发现
图1基因组到蛋白质组全景图谱的构建流程与关键发现
2.核苷酸分辨率的基因组-蛋白质组图谱构建
蛋白质组学分析共鉴定出6476个蛋白质数量性状位点(pQTLs),它们控制着923个蛋白质的丰度,其中1650个pQTLs(占比25.5%)可精细定位到单个核苷酸多态性,达到“核苷酸分辨率”(图1G-K);该图谱对蛋白质丰度整体方差的解释率达22.8%,与酵母mRNA水平的数量性状位点(eQTL)图谱解释力相当,且通过CRISPR基因编辑可直接验证单个pQTN的调控效应,证明图谱的准确性与可靠性(图2A-D)。
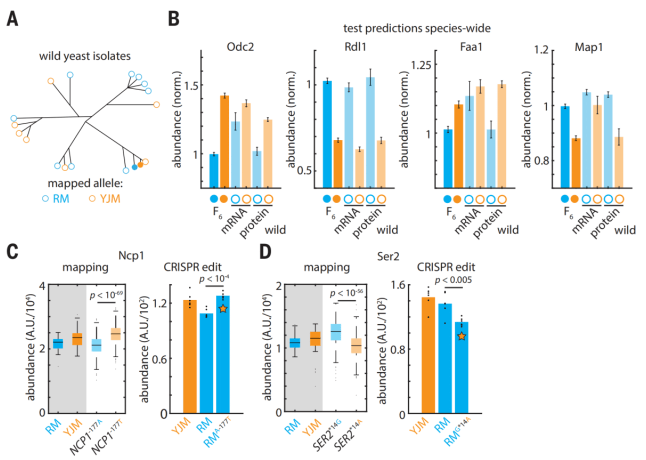 图2 变异-分子图谱揭示蛋白质水平的调控机制
图2 变异-分子图谱揭示蛋白质水平的调控机制
3.顺式与反式调控变异的关键特征
研究发现98%的pQTLs属于反式关联(即调控位点与靶蛋白编码基因距离>1kb),仅2%为顺式关联;尽管单个顺式pQTL的效应量更大,但反式pQTLs的累积效应更强;更重要的是,反式调控并非传统认知中的转录因子,而是代谢酶(如肌苷单磷酸脱氢酶IMD2)或膜转运体(如尿嘧啶转运体FUR4);且编码变异的效应显著大于非编码变异,因其可同时影响调控蛋白的丰度与功能,而非编码变异仅能影响丰度(图3)。
 图3 所有数量性状基因座(pQTL)的基因组定位
图3 所有数量性状基因座(pQTL)的基因组定位
4.自然选择对蛋白质组的塑造
通过pQTL统计检验发现,pQTLs对蛋白丰度的调控效应高度相关,表明亲本菌株经历了两种选择压力——定向选择(424个蛋白的子代丰度分布比预期更窄,说明选择倾向于特定表达水平)和稳定选择(286个蛋白的子代变异性比预期更高,说明选择维持多样性);生态位分析显示,73个pQTL等位基因在发酵环境中显著富集,24个在人类相关环境中富集,证明蛋白质组变异与菌株的生态适应性直接相关(图4B-F)。
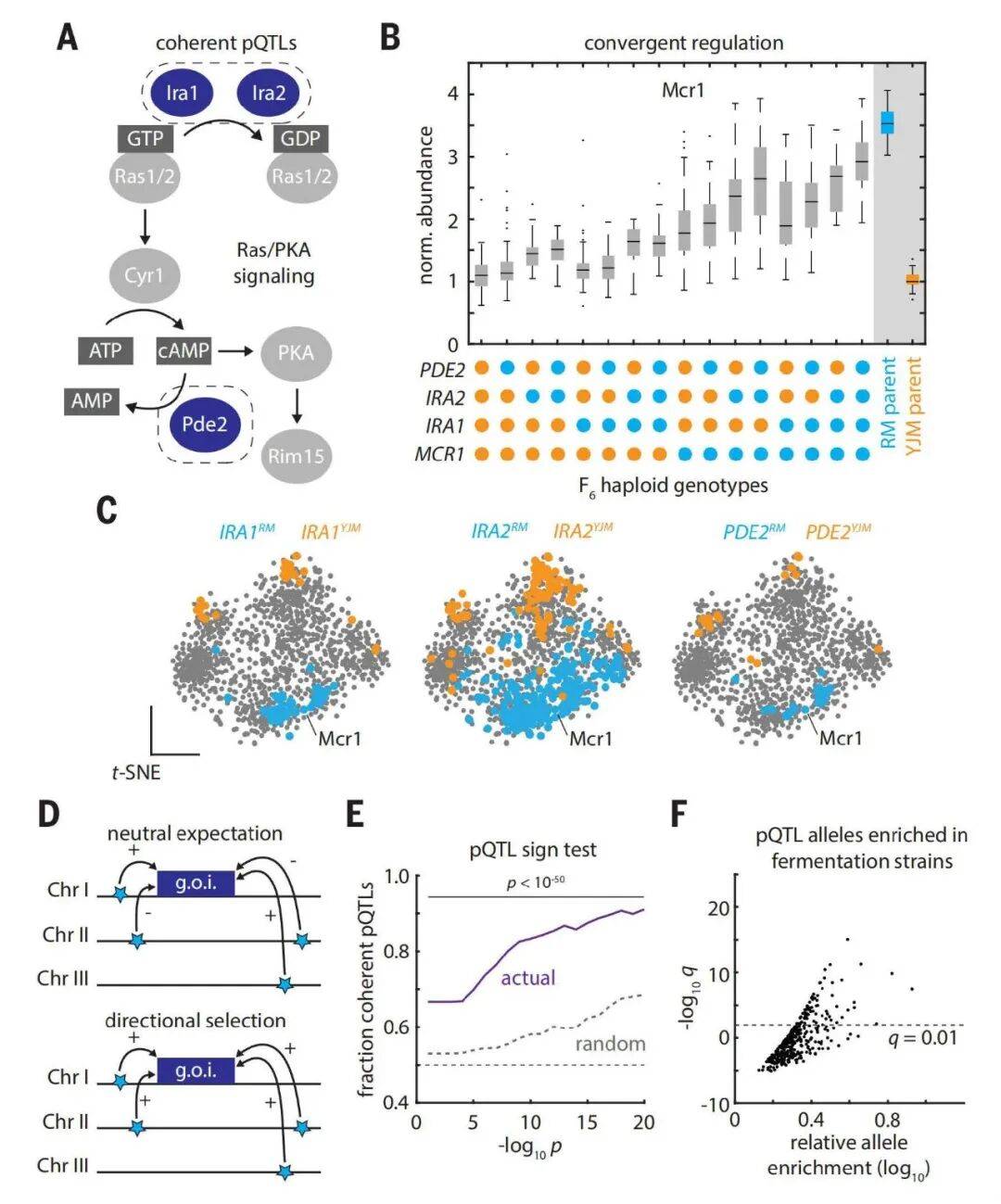 图4 多基因适应反映了蛋白质丰度的自然选择
图4 多基因适应反映了蛋白质丰度的自然选择
5.跨环境的预测能力与功能验证
尽管图谱是在标准无胁迫条件下构建的,但其中的pQTLs可跨环境预测遗传变异的表型效应——例如,ERG11基因的两个变异(调控型C122014C与错义型Lys433Asn)可通过图谱预测其对氟康唑(抗真菌药物)敏感性的影响,且CRISPR验证显示两者协同降低敏感性(图5A-F);此外,跨环境分析发现,78%的胁迫抗性QTL(如抗药物、抗金属胁迫)与pQTL共定位,远高于胁迫条件间QTL的共定位率(48%),证明蛋白质组是“潜在分子层”,可提前揭示遗传变异在未发生胁迫时的表型潜力(图6J-L)。
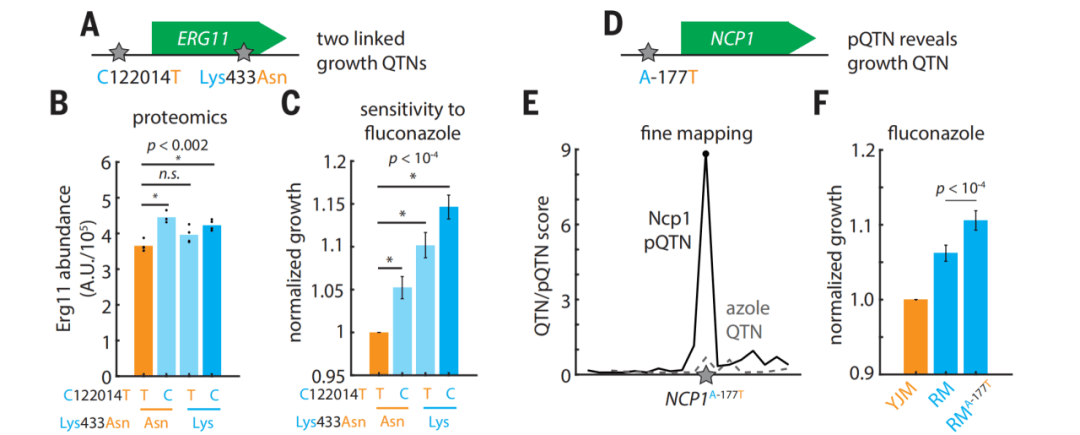 图5 变体-蛋白质图谱中嵌入的隐性适应度效应
图5 变体-蛋白质图谱中嵌入的隐性适应度效应
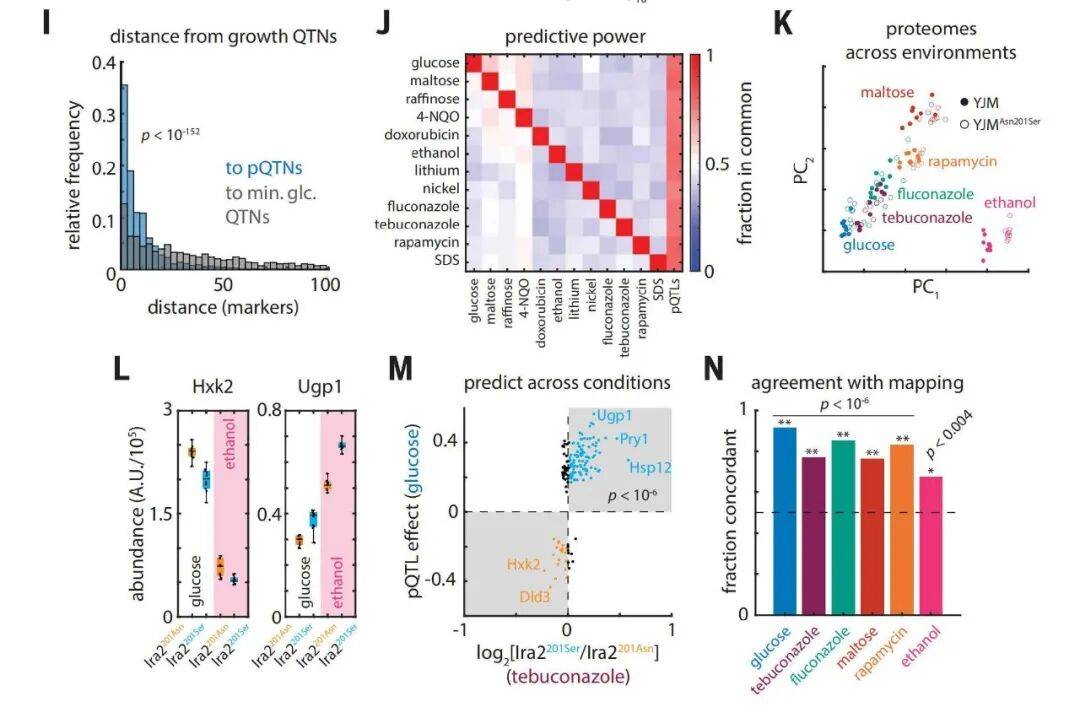 图6 蛋白质组鉴定出定量性状背后的因果变异
图6 蛋白质组鉴定出定量性状背后的因果变异
文章小结
本研究构建了首个自然变异下的核苷酸分辨率基因组-蛋白质组图谱:该图谱鉴定出6476个基因型-蛋白质关联(pQTL),其中1650个可定位到单个多态性位点(pQTN),揭示了顺式和反式调控变体(尤其是代谢酶/转运体来源的反式调控热点)对蛋白质组多样性的驱动作用,证实自然选择塑造蛋白质组进化,并发现该图谱可跨环境预测遗传变异的表型效应,为理解遗传适应机制及预测遗传变体功能提供了关键框架。
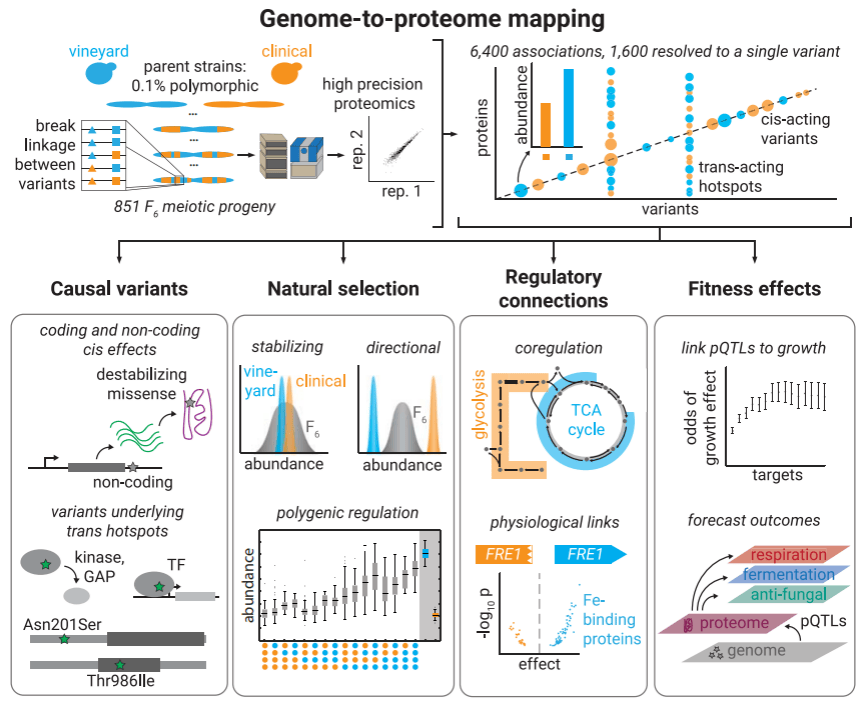 图7 基因组到蛋白质组全景图谱
图7 基因组到蛋白质组全景图谱
拜谱小结
该研究构建的核苷酸分辨率基因组-蛋白质组图谱,揭示了自然变异通过顺反调控、多基因适应驱动蛋白质组多样性的机制,为从基因组到复杂性状的分子关联研究提供了关键框架。拜谱生物作为一家国内领先的多组学服务公司,可提供完善成熟的蛋白质组学、修饰组学、代谢组学、转录组学等多组学产品技术服务体系,整合多组学数据进行深入挖掘分析,全面解析机制机理等,助力高分文章发表。欢迎咨询!
参考文献:
Jakobson CM, Hartl J, Trébulle P,et al. A genome-to-proteome map reveals how natural variants drive proteome diversity and shape fitness. Science. 2025;390(6769):eadu3198. doi:10.1126/science.adu3198