

前言
细胞内部区室化是人类细胞结构的基本特征,以细胞器为单位的细胞区室化对于细胞空间构成、行使独立的细胞功能、隔离有害化学物质等细胞生物过程至关重要。细胞区室化通过细胞组分动态的亚细胞定位控制细胞生物学过程,从而参与细胞与环境的互作。因此,认识细胞器的分子组成,特别是蛋白质的组成,以及这种组成如何在细胞状态之间重塑仍然是人类细胞生物学的一个主要目标。过往基于成像或质谱表征细胞器蛋白质组的研究方法仍存在鉴定深度低、操作繁琐、只能分析少数细胞器等局限性。
2025年1月2日,Cell期刊在线发表了扎克伯格Biohub研究所Manuel D. Leonetti团队的文章 “Global organelle profiling reveals subcellular localization and remodeling at proteome scale”。本研究基于细胞器免疫捕获与质谱联用开发了一种高分辨率绘制亚细胞级蛋白质组图谱的工作流程。研究人员利用此流程获得了人类细胞全区室细胞器蛋白质组图谱,注释了 7,600 多种蛋白质的亚细胞定位,并定义了细胞空间网络,揭示了细胞区室之间的关联。该方法可以用于全面分析细胞扰动期间的蛋白质组重塑。作者通过表征 HCoV-OC43 病毒感染后的细胞景观,表明亚细胞重塑的蛋白质组分析为阐明细胞生物学过程提供了关键见解。本研究数据集可以在organelles.czbiohub.org开放获取。

研究结果
01、基于细胞器免疫捕获的全细胞区室蛋白质组检测流程
简单来说,该流程就是基于免疫捕获的方法获得细胞内所有区室(compartments),然后通过质谱的方法对富集的区室蛋白质组进行表征。作者给所有的区室分配了特异性marker蛋白,包括有细胞质膜的细胞器、无膜的区室以及胞质聚集体,共37个marker蛋白和19个细胞区室(图1D)。首先是通过CRISPR-Cas9给每个细胞器的marker基因添加了表位标签(作为“钓饵”):linker-GFP-3xHA(图1A)。通过流式细胞分选,获得表达细胞器marker蛋白的细胞文库(图1B)。然后在天然温和条件下进行细胞裂解,保持细胞器完整性,用免疫沉淀捕获带有标签的细胞区室,用分段离心的方法获得那些未标记的细胞核、膜结合细胞器以及胞浆部分(图1CD)。质谱分析的蛋白质组通过试剂对照和IgG对照,选取差异倍数2倍,P≤0.01的蛋白作为细胞区室富集的蛋白质组。
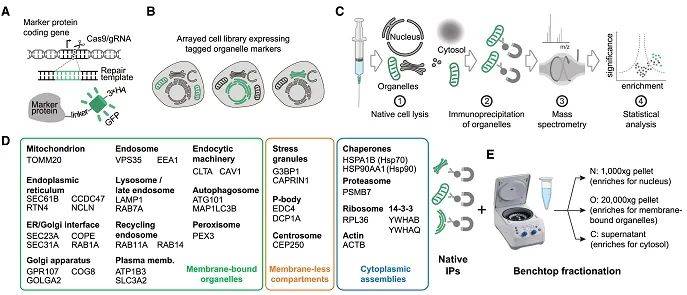 图1 本研究所采用的全细胞区室蛋白质组检测流程
图1 本研究所采用的全细胞区室蛋白质组检测流程
(图源:Hein et al., Cell, 2025)
02、绘制亚细胞级别的人类蛋白质组图谱
基于上述方法作者获得HEK293T细胞的全细胞区室蛋白质组数据集,共钓取8,538个蛋白(图2A)。蛋白质Pearson相关性分析表明同一细胞器内的蛋白高度相关联,部分细胞内直接互作的细胞器的蛋白质也呈现中度的关联,比如内质网和线粒体蛋白(图2B)。另外,细胞内互作蛋白也能呈现高度相关性或共富集。
为了进一步挖掘富集蛋白组的特征,作者参考了单细胞转录组降维的思想,采用K-最邻近(K-NN)算法对高维的全细胞区室蛋白质组数据矩阵进行降维分析。结合GO数据库亚细胞定位富集分析,最后利用正则化为每个蛋白质分配其相邻蛋白中最具代表性的标记(图2C)。经过聚类分析,作者获得了HEK293T细胞二维的亚细胞蛋白质图谱UMAP图(图2D)。该图谱注释了20个独立的亚细胞区室,区室的分布符合已知的生物学过程,比如内质网、高尔基体、内体、细胞膜蛋白质的分布关系与细胞分泌系统一致。
基于前期公布的亚细胞蛋白质组数据集,作者构建了机器学习分类器来验证基于降维图形化注释结果的准确性,结果显示AUC≥0.93。作者还基于最全面的线粒体蛋白质组数据库评价了本研究的注释召回率在所有已公布数据集中最高(77%)。总之作者通过与过往亚细胞蛋白质组数据进行比较分析,验证了本研究注释结果的客观真实性。
 图2 人类亚细胞蛋白质组图谱的图形化呈现
图2 人类亚细胞蛋白质组图谱的图形化呈现
(源自:Hein et al., Cell, 2025)
03、亚细胞蛋白网络定义功能特征并量化细胞器连接
亚细胞蛋白质图谱中的k-NN map是某个蛋白与其邻近的20个蛋白的网络图,从k-NN map中能挖掘蛋白功能信息。作者以WASHC5为例进行了说明,这是一个负责内体-高尔基体-溶酶体系统分子转运的调节因子,k-NN map显示WASHC5定位于内体-高尔基体-溶酶体的中心交界处(图3A)。与其相连的蛋白分别是Retromer和Commander复合物(图3B),也反映了WASHC5与Retromer或Commander复合物关联,从而运输不同分子的功能。总而言之,该示例表明了如何挖掘 k-NN map的详细结构以进行功能探索。
细胞器间通讯是细胞生理学的重要组成部分。作者利用不同亚细胞cluster之间的k-NN连接数量即连接密度对亚细胞区室之间的联系进行度量(图3C)。为了进一步注释细胞器之间的关联,作者在k-NN map中鉴定了那些界面蛋白,并认为这些界面蛋白是负责连接不同细胞器的关键蛋白(图3D,E)。通过这种方法,作者准确识别了许多专门用于囊泡系统内界面运输或调节的蛋白质(图3F)。
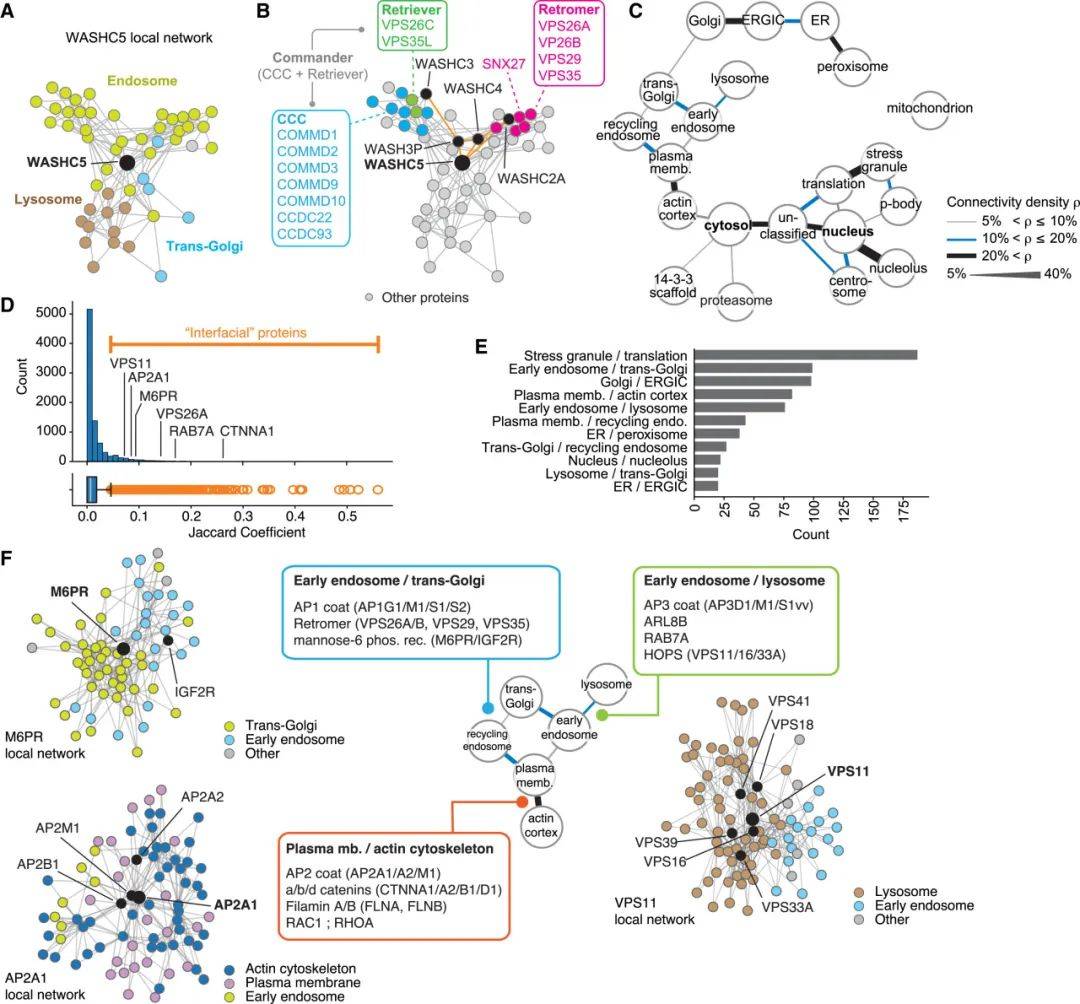 图3 蛋白质空间网络识别协同功能蛋白质和细胞器界面
图3 蛋白质空间网络识别协同功能蛋白质和细胞器界面
(源自:Hein et al., Cell, 2025)
04、HCoV-OC43病毒感染导致的泛细胞重塑(pan-cellular remodeling)
为了获得外界扰动对细胞内环境重塑的全面性认识,作者基于上述方法研究了HCoV-OC43病毒感染条件下的全局细胞器蛋白质组。感染后48h取样,分析了28个免疫捕获细胞器和3个分段离心的蛋白质组。将亚细胞蛋白质组转换成k-NN map,再通过aligned-UMAP算法将感染和非感染亚细胞蛋白质组整合(图4A)。每个蛋白赋予其亚细胞重塑得分来衡量感染后蛋白的亚细胞重定位。根据亚细胞重塑得分获得感染相关的633个蛋白,UMAP图和桑基图显示这633个蛋白的亚细胞环境发生显著的改变(图4D-E),表明病毒感染导致了细胞内环境的全局性重塑。其中ERGIC、中心体、内质网、高尔基体中呈现最高的亚细胞重塑(图4F):这与已知的冠状病毒在内质网/高尔基体界面复制和组装的细胞生物学相匹配。活细胞共聚焦显微镜成像也表明高尔基体、内质网和围观网络在病毒感染后发生明星的亚细胞重塑(图4G-H)
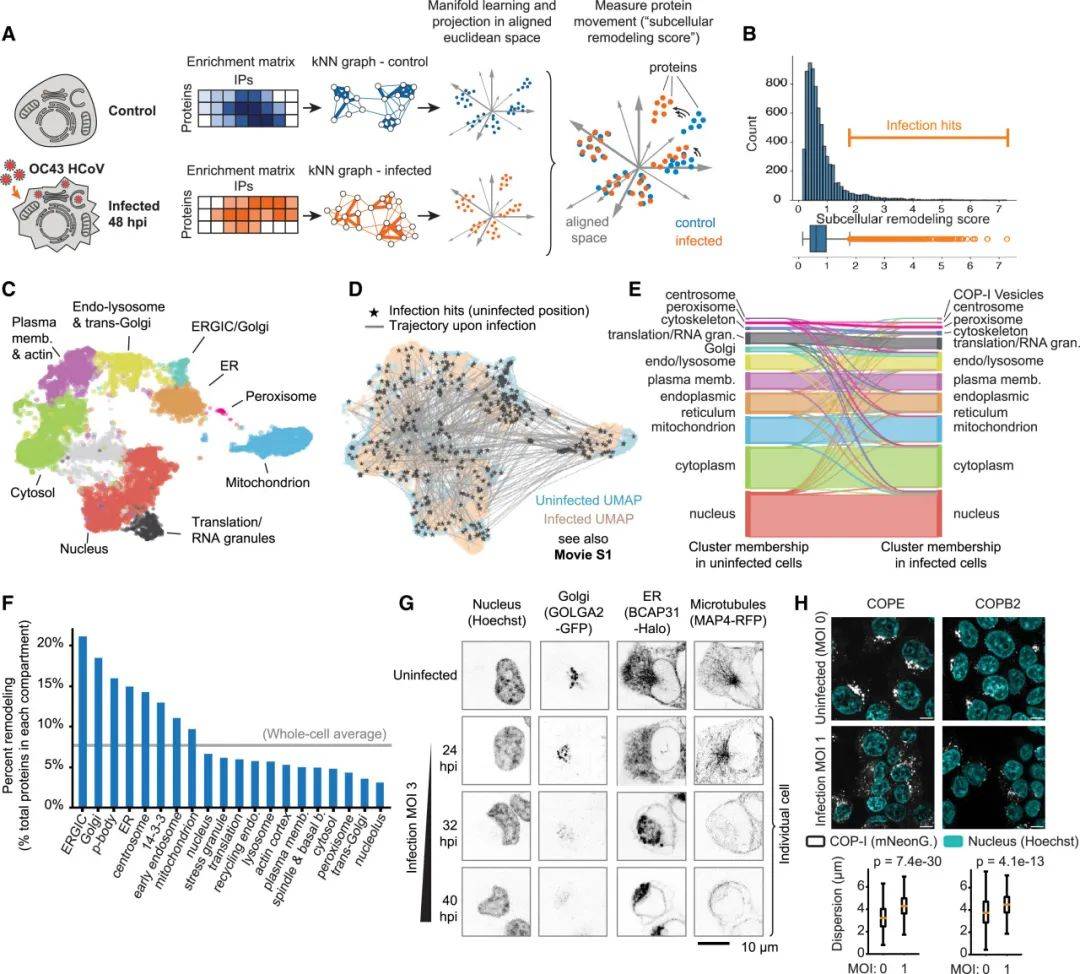 图4 HCoV-OC43感染引起的泛细胞重塑
图4 HCoV-OC43感染引起的泛细胞重塑
(源自:Hein et al., Cell, 2025)
05、亚细胞重塑重新定义病毒感染的细胞生物学过程
作者比较了亚细胞蛋白质组和基因表达水平的蛋白质组或转录组,发现二者只有少部分的交集,表明了功能调节的正交模式,即许多蛋白质在其空间环境水平上受到动态调节,而不是受到细胞丰度变化的调节。亚细胞重塑蛋白质功能富集分析揭示了病毒感染相关的糖胺聚糖代谢、铁死亡、自噬和细胞衰老通路(图5A-B)。作者进一步验证了铁死亡在促进病毒感染过程中的作用(图5C-D)。有意思的是,作者同k-NN map分析发现铁死亡通路上的蛋白NCOA4亚细胞环境发生显著变化,病毒感染后NCOA4相邻的蛋白变为激活、延长的自噬体相关因子,揭示了铁蛋白自噬激活在 OC43 感染的细胞生物学中的作用(图5F)。
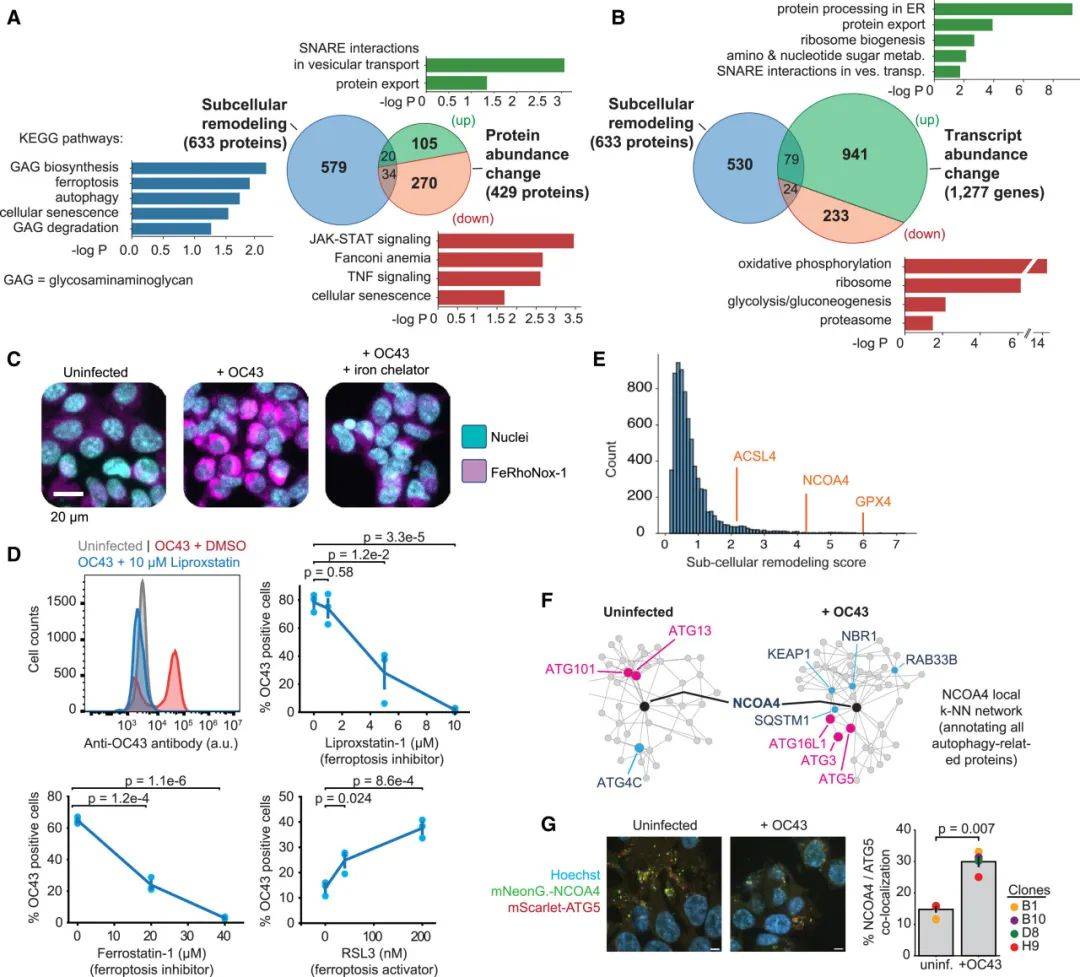 图4 HCoV-OC43感染引起非丰度变化的细胞反应
图4 HCoV-OC43感染引起非丰度变化的细胞反应
(源自:Hein et al., Cell, 2025)
小结
综上所述, 本研究创建了一套亚细胞蛋白质组表征和动力学分析的工作流程,包含了实验部分和数据分析。首先基于全细胞器的marker蛋白,利用免疫捕获收集全细胞器,并进行质谱检测,获得亚细胞蛋白质组数据矩阵;然后开发了基于图形的数据分析流程,用来识别蛋白质的功能关系和亚细胞定位注释,获得亚细胞蛋白质组图谱;最后应用该工作流程研究了细胞在外界扰动下的亚细胞蛋白质组动态特征和亚细胞环境重塑。以HCoV-OC43病毒感染细胞为例,本工作流程揭示了非蛋白丰度改变的亚细胞重塑细胞反应,推动了对病毒感染细胞生物学的认识。
拜谱小结
从亚细胞重塑的角度理解细胞与环境的互作,为进一步认识疾病相关的病理和生理机制,发现疾病治疗靶点提供了新的科学依据。随着技术的进步,从bulk蛋白质组到单细胞蛋白质组,再到如今的亚细胞蛋白质组,分辨率越来越高,促进了从不同维度和层次下识别蛋白质的动态特征。Bulk蛋白质组识别组织或细胞样本平均的蛋白表达水平,单细胞蛋白质组则更关注的是样本中的细胞异质性,那么亚细胞蛋白质组则告诉我们细胞内环境蛋白分子的空间变化信息,即亚细胞定位的重塑。
拜谱生物作为国内领先的多组学公司,可提供完善成熟的蛋白组学、代谢组学、转录组学等多组学产品技术服务体系。其中本司开发了高深度的DIA-蛋白质组、极微量蛋白质组、单细胞蛋白质组和空间蛋白质组,可助力研究人员开展不同尺度下的蛋白质组学研究,揭示蛋白分子动态规律和微观空间特征。DIA-蛋白质组适用于常规样本,检测深度更高、重复性更好、灵敏度更高;极微量蛋白质组适用于原代细胞、卵母细胞、临床穿刺样本等微量样本的蛋白质组学研究;单细胞蛋白质组通常结合单细胞转录组进行研究,不仅能对单细胞转录组的结果进行验证,还能获得蛋白质层面的表达信息;空间蛋白质组可实现对病理切片中特定空间位置组织微环境在成像及蛋白表达等层面进行系统的定位、定性和定量分析,揭示组织微环境高度异质性、不同细胞类型的空间分布。欢迎咨询!
参考文献:
Hein MY, Peng D, Todorova V, et al. Global organelle profiling reveals subcellular localization and remodeling at proteome scale. Cell. Published online December 26, 2024. doi:10.1016/j.cell.2024.11.028