

机器学习是一种使计算机系统能够从数据中学习并做出预测或决策的技术。它是人工智能(AI)的一个重要分支,主要依赖于统计学、优化理论和计算机科学的方法。机器学习在肿瘤研究中扮演着越来越重要的角色,在肿瘤生物标志物、分子分型、药物发现和开发等领域中广泛应用。比如利用机器学习分析肿瘤基因组数据,识别与癌症相关的基因变异和突变,开发肿瘤早期诊断的预测模型;在药物发现阶段,机器学习可以预测小分子药物与靶点的结合能力,加速药物筛选过程。
近期,来自哈佛医学院的研究人员开发了一种基于AKT和EZH2双重抑制剂的三阴乳腺癌治疗方法,并利用机器学习筛选和构建了疗效敏感性预测模型,相关成果发表在Nature期刊上。

三阴性乳腺癌(TNBC)是最具侵袭性的乳腺癌亚型,复发率最高。晚期TNBC的主要治疗标准是进行全身化疗联合免疫治疗,或单独全身化疗;然而,治疗反应通常持续时间很短。因此,迫切需要开发更有效的治疗方法。
PI3K信号通路的组成部分是潜在的治疗靶点,因为在超过70%的TNBC病人中,PIK3CA、AKT1或PTEN均发生了改变。然而,与激素受体阳性乳腺癌亚型相比,目前尚不清楚TNBC是否会对PI3K通路抑制剂产生反应,以及会产生何种反应。
研究人员利用TNBC细胞系和小鼠模型测试了AKT抑制剂和组蛋白甲基转移酶EZH2抑制剂联合使用能有效杀死肿瘤细胞,有趣的是单独使用其中一种抑制剂对肿瘤没有效果。随后作者利用转录组测序和体内荧光成像等技术揭示了EZH2和AKT抑制剂可驱动TNBC细胞分化,促进其转变成管腔样(luminal-like)细胞状态,这是双重抑制剂抗肿瘤作用的关键因素。
研究人员聚焦管腔细胞分化的关键调节因子GATA3研究了双重抑制剂的作用机制。研究发现EZH2抑制能打开了增强子序列上的染色质从而调节GATA3的表达。AKT的抑制能阻碍FOXO1的磷酸化,并触发FOXO1在增强子和启动子序列上的结合。一旦细胞分化,EZH2和AKT抑制剂就能通过劫持退化过程中的信号分子来驱动细胞凋亡。进一步,这一过程通过诱导IL-6—JAK1—AT3通路,触发促凋亡蛋白BMF的表达来实现的。
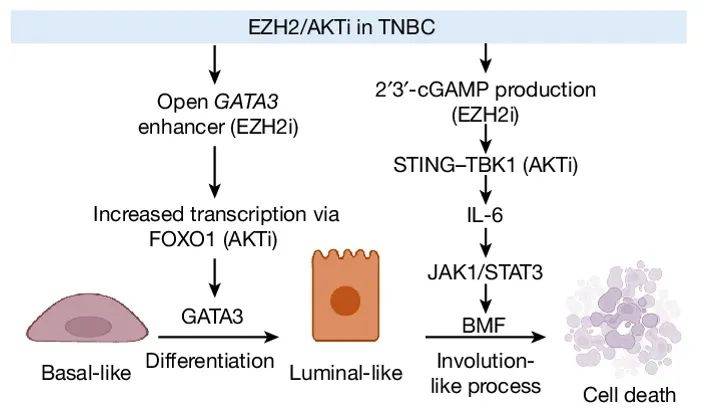 EZHE/AKT双重抑制剂治疗TNBC的作用机制模式图
EZHE/AKT双重抑制剂治疗TNBC的作用机制模式图
(图源Schade AE, et al., Nature, 2024)
研究发现EZH2和AKT抑制剂治疗TNBC细胞系呈现敏感型和耐药型,于是研究人员基于机器学习开发了药物治疗反应的预测模型。首先选择了17个细胞系(包含10个敏感细胞系和7个耐药细胞系)的转录组数据集,根据敏感型和耐药型的差异表达基因集(5组变量)作为训练集,随后选择随机森林和支持向量机算法构建预测模型并利用留一交叉验证策略(leave-one-out cross-validation)验证模型预测性能。根据预测性能准确性,研究人员选择差异倍数大于5倍的差异表达基因集作为变量和随机森林算法作为最优的预测模型。最后作者利用TCGA中TBNC组织样本的转录组数据库作为独立验证集对预测模型进行了验证,预测结果显示55%的样本对于EZH2/AKT双重抑制剂治疗是敏感的,这一发现与本研究中细胞系的敏感率(60%)相似。
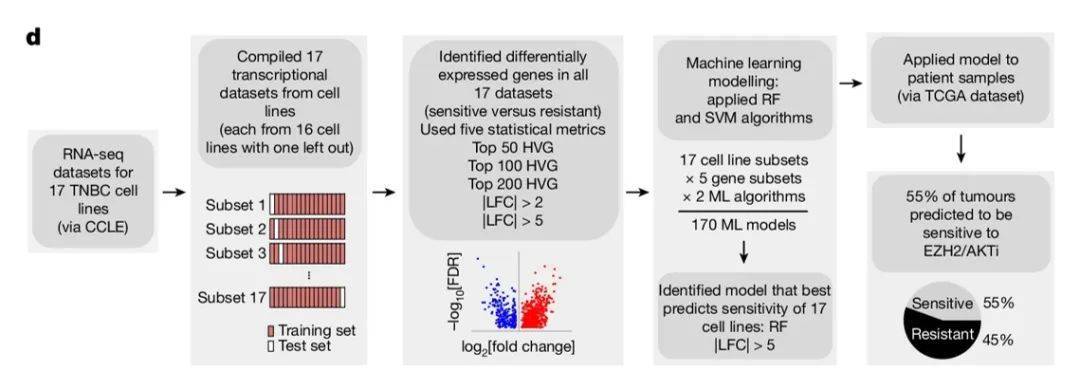 机器学习模型训练和预测评估工作流程示意图
机器学习模型训练和预测评估工作流程示意图
(图源Schade AE, et al., Nature, 2024)
综上,本研究为这种高度侵袭性肿瘤类型确定了一种有希望的治疗策略,并说明了解除管制的表观遗传酶如何将肿瘤与致癌脆弱性隔离开来。这些研究还揭示了发育组织特异性细胞死亡途径如何被用于治疗益处。
拜谱小结
生物标志物的发现和验证对于疾病的早期诊断、治疗监测和预后评估至关重要。机器学习可以从大量的基因组学、蛋白质组学、代谢组学和其他类型的生物数据中挖掘出潜在的生物标志物,并通过算法筛选出与疾病状态最相关的生物分子或特征。使用机器学习分类算法对生物标志物进行分类,区分疾病状态和健康状态,进而通过建立机器学习模型进行疾病的诊断、预测疾病的发展和治疗反应或患者预后。
拜谱生物拜作为国内领先的多组学公司,可提供完善成熟的蛋白组学、代谢组学、转录组学等多组学产品技术服务体系。拜谱生物建立了完善的基于蛋白质组学、代谢组学的机器学习分析流程,可实现随机森林、极端梯度提升(Xgboost)、支持向量机(SVM)、朴素贝叶斯法(Naive Bayes, NB)、K-近邻法(K-Nearest Neighbors,KNN)、LASSO逻辑回归等经典机器学习算法分析,同时可提供ROC、PR、混淆矩阵、生存分析等临床性能分析,为临床大队列和生物标志物研究提供了丰富的选择。
参考文献:
Schade AE, Perurena N, Yang Y, et al. AKT and EZH2 inhibitors kill TNBCs by hijacking mechanisms of involution. Nature. Published online October 9, 2024. doi:10.1038/s41586-024-08031-6B1